pyrin基因序列的调取
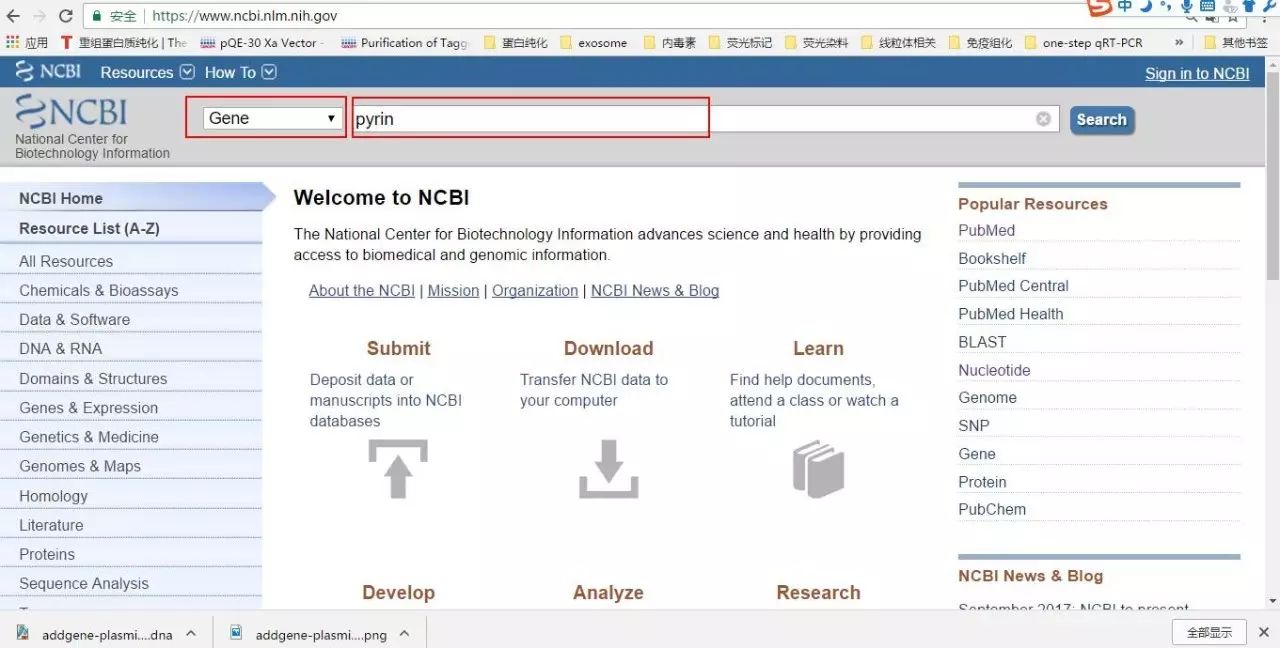
进入NCBI数据库(http://www.ncbi.nlm.nih.gov),
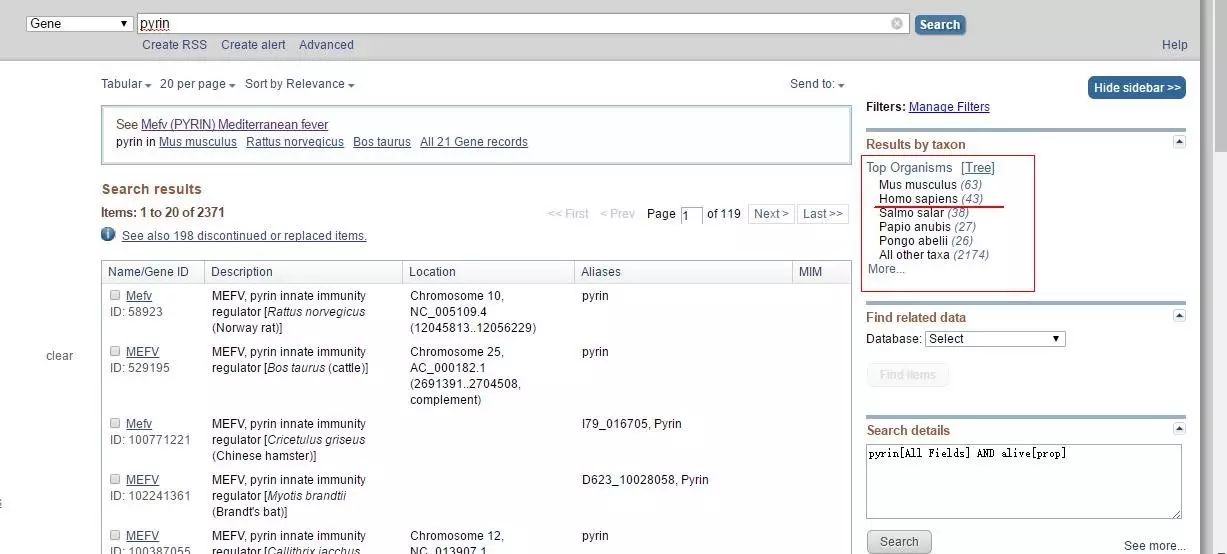
选择pyrin种属来源,如人
选择基因亚型,需查阅文献(根据实验需求,并查阅文献发现pyrin亚型1与研究目的一致)
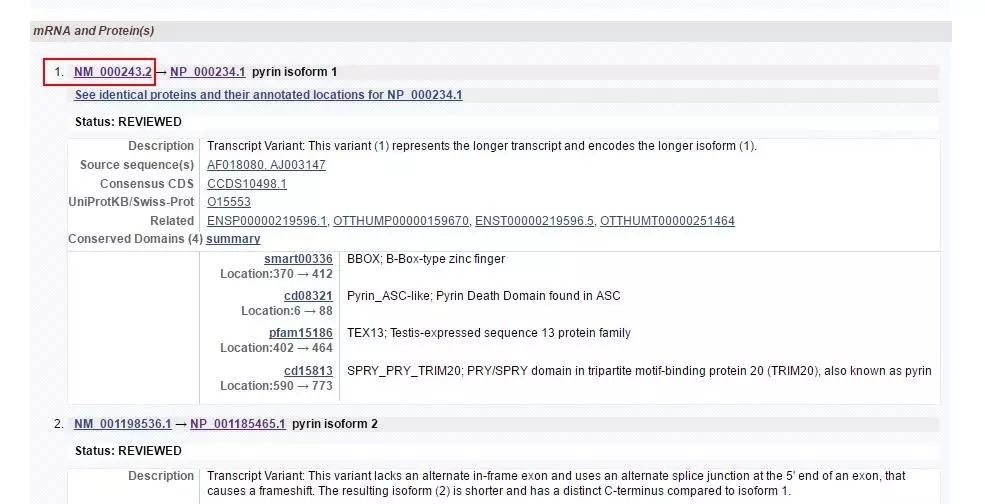
点击NM 000243.2,查找到基因pyrin的CDS区,
2F
克隆方法的选择
粘性末端克隆法
该方法需要在目的基因pyrin引物的5’端加上酶切位点,方法如下:
1. 酶切位点选择
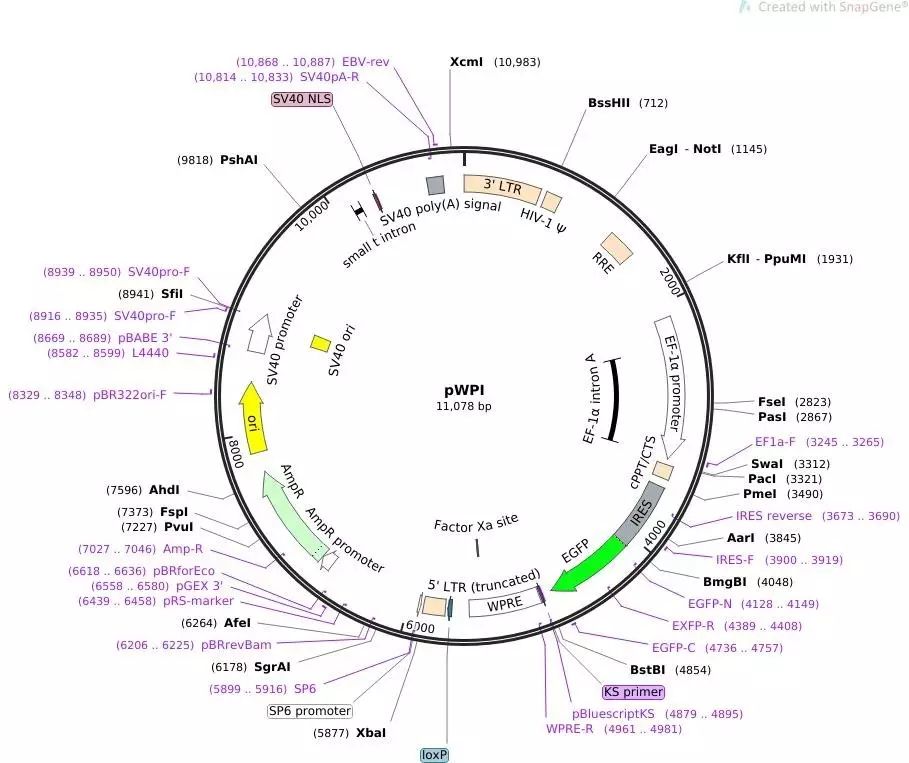
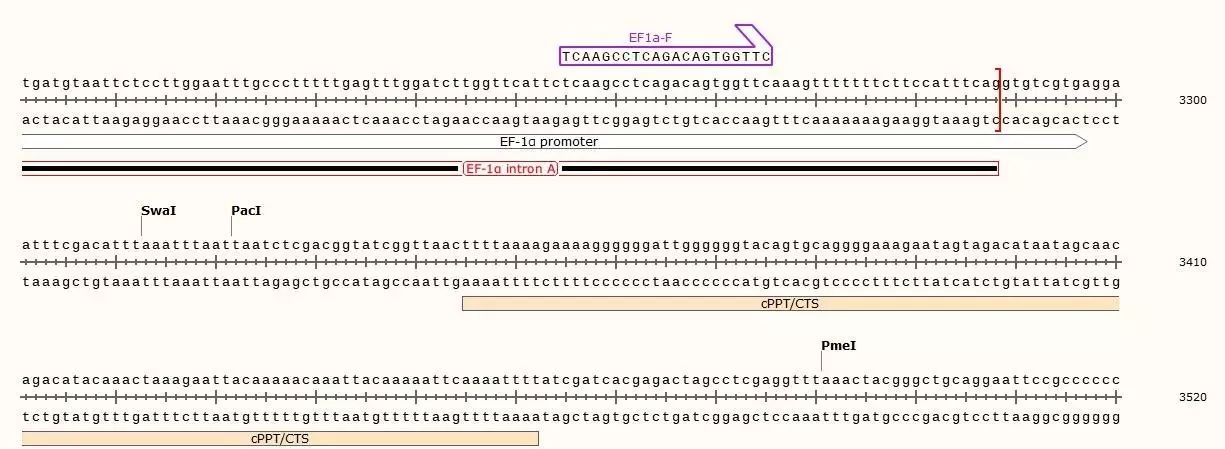
1)pWPI-eGFP质粒中带有启动子EF-1α(与CMV启动子类似),双酶切可选择SwaⅠ与PmeⅠ或PacⅠ与PmeⅠ。注意不要选SwaⅠ与PacⅠ,因为双酶切时两个酶切位点的距离应相差在9bp以上。在这里,我们选用PacⅠ与PmeⅠ双酶切距离说明。
2)双酶切时,需要在酶切位点前面加上保护碱基,以保证最佳的酶切效率。选取酶切效率最高的保护碱基。
2. pyrin基因的引物设计
以PacⅠ与PmeⅠ双酶切为例,pyrin基因的上下游引物为
3. 设计好引物后,PCR扩增pyrin基因,跑胶回收PCR产物,PacⅠ与PmeⅠ双酶切pWPI质粒和pyrin基因,回收酶切产物,连接转化即可。
4. 挑取单克隆,验证,获得阳性克隆。
粘性末端克隆获得的重组质粒可用于瞬转,研究基因功能。但是,要长时间稳定过表达基因,就需要慢病毒包装,此时pWPI-eGFP中的cPPT/CTS需要保留,那么PmeⅠ就不适合进行酶切载体。此时就需要另外一种克隆方法——一步法克隆。
一步法克隆法
该方法需要在pyrin基因引物的5’端添加与pWPI载体末端相同的重叠序列,具体做法如下:
1. 酶切位点的选择
根据慢病毒包装的要求,pyrin基因需要插入在cPPT/CTS位点前面,此时载体的线性化可以通过SwaⅠ或PacⅠ单酶切实现。
2. pyrin基因的引物设计
1)重叠序列大小为15-20bp,酶切位点不计算在内;2)以PacⅠ单酶切为例,pyrin基因的上下游引物为:
3. 设计好引物后,PCR扩增pyrin基因,胶回收产物与载体酶切回收产物发生重组反应。
4 挑取单克隆,验证,获得阳性克隆。
3F
质粒构建成功后,转染细胞,观察是否表达
若进行稳转,后续还需使用该重组质粒包装慢病毒。注意事项
1、可通过GFP蛋白的表达情况,观察转染效率。若转染效率较低,可通过抗生素筛选,提高整合基因到基因组的细胞的纯度。因pWPI-eGFP质粒不带有哺乳动物细胞药物筛选基因,因此可以通过流式细胞术筛选,获得带有GFP阳性的细胞,使整合基因到基因组的细胞纯度提高。
2、还可以通过有限稀释法获得单个阳性克隆的细胞,避免在细胞传代过程中,由于细胞不断分裂,过表达的效果不断减弱造成的表型不明显现象的发生。
3、本案例较为特殊,对于其他过表达载体构建或慢病毒包装而言,一步法克隆方法与粘性末端克隆方法或许均可使用。