
长期以来,化学家们一直梦想着全面了解和掌握生命的化学工具--蛋白质。现在,这个梦想已经触手可及。Demis Hassabis 和 John M. Jumper 成功地利用人工智能预测了几乎所有已知蛋白质的结构。David Baker 学会了如何掌握生命的组成元素,并创造出了全新的蛋白质。他们的发现潜力巨大。
生命的化学是如何如此充满活力的?答案是蛋白质的存在,可以将其描述为卓越的化学工具。它们通常由 20 种氨基酸组成,这些氨基酸可以以无数种方式组合在一起。利用储存在 DNA 中的信息作为蓝图,氨基酸在我们的细胞中被连接成长长的链。
然后蛋白质的魔法就发生了:氨基酸链扭曲并折叠成独特的三维结构(图 1),这种结构有时是独一无二的。这种结构赋予了蛋白质功能。其中一些成为化学建筑材料,可以创建肌肉、角或羽毛,而另一些则可能成为激素或抗体。许多蛋白质形成酶,这些酶以惊人的精度驱动生命的化学反应。位于细胞表面的蛋白质也很重要,它们充当细胞与其周围环境之间的通信渠道。
(来源:Johan Jarnestad/The Royal Swedish Academy of Sciences)
生命的化学构建——这 20 种氨基酸——所蕴含的潜力之大,无论怎样强调都不为过。2024 年诺贝尔化学奖就是要在一个全新的水平上理解和掌握它们。一半奖项授予了 Demis Hassabis 和 John Jumper,他们利用人工智能成功地解决了化学家们 50 多年来一直在努力解决的问题:从氨基酸序列预测蛋白质的三维结构。这使得他们能够预测几乎所有已知的 2 亿种蛋白质的结构。另一半奖项授予了 David Baker。他开发了计算机化方法,实现了许多人认为不可能的事情:创造以前不存在的蛋白质,并且这些蛋白质在许多情况下具有全新的功能。
2024 年诺贝尔化学奖表彰的是两项不同的发现,但正如你将看到的,它们之间有着密切的联系。要了解今年的获奖者所克服的挑战,我们必须回顾一下现代生物化学的黎明时期。
蛋白质的第一批模糊图片
自 19 世纪以来,化学家们就知道蛋白质对生命过程很重要,但直到 20 世纪 50 年代,化学工具才足够精确,研究人员才开始更详细地探索蛋白质。剑桥大学的研究人员 John Kendrew 和 Max Perutz 在该十年的末期取得了突破性发现,他们成功地使用了一种称为 X 射线晶体学的方法,展示了蛋白质的第一个三维模型。他们因这一发现获得了 1962 年诺贝尔化学奖。
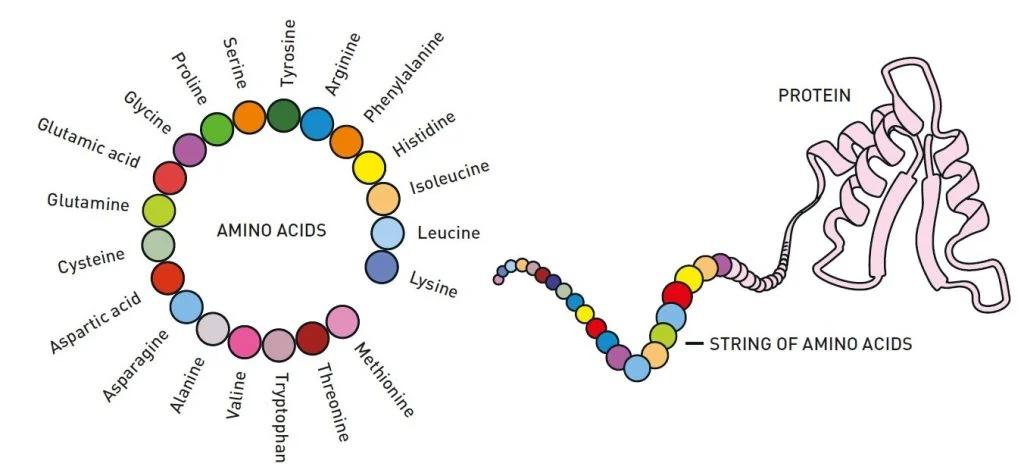
图 1. 蛋白质可以由几十个到几千个氨基酸组成。一串串氨基酸折叠成三维结构,对蛋白质的功能起着决定性作用。(来源:ohan Jarnestad)
随后,研究人员主要使用 X 射线晶体学(通常需要付出大量努力)成功制作了约 20 万种不同蛋白质的图像,这为 2024 年诺贝尔化学奖奠定了基础。
一个谜:蛋白质是如何找到其独特结构的?
美国科学家 Christian Anfinsen 早在 1961 年就做出了另一项早期发现。他使用各种化学技巧,设法使现有蛋白质展开,然后又折叠起来。有趣的是,蛋白质每次都恢复到完全相同的形状。他得出结论,蛋白质的三维结构完全由蛋白质中的氨基酸序列决定。这使他获得了 1972 年诺贝尔化学奖。
然而,Anfinsen 的逻辑中存在一个悖论,另一位美国人 Cyrus Levinthal 在 1969 年指出了这一点。他计算出,即使蛋白质只由 100 个氨基酸组成,理论上蛋白质也可以采取至少 10^47 种不同的三维结构。如果氨基酸链随机折叠,那么找到正确的蛋白质结构需要比宇宙的年龄还要长的时间。在细胞中,这只需要几毫秒。那么氨基酸链实际上是是如何折叠的呢?
Anfinsen 的发现和 Levinthal 的悖论表明,折叠是一个预先确定的过程。而且——重要的是——蛋白质折叠的所有信息都必须存在于氨基酸序列中。
迎接生物化学的巨大挑战
上述见解导致了另一个决定性的认识——如果化学家们知道蛋白质的氨基酸序列,他们应该能够预测蛋白质的三维结构。这是一个令人兴奋的想法。如果他们成功了,他们就不再需要使用繁琐的 X 射线晶体学,并可以节省大量时间。他们还将能够为所有无法使用 X 射线晶体学获得的蛋白质生成结构。
这些逻辑结论向生物化学的伟大挑战发出了挑战:预测问题。为了促进该领域的更快发展,1994 年研究人员启动了一个名为“蛋白质结构预测关键评估”(CASP)的项目,该项目发展成了一场竞赛。每两年,来自世界各地的研究人员都会获得刚刚确定结构的蛋白质的氨基酸序列。然而,参赛者无法获知这些结构。挑战是根据已知的氨基酸序列预测蛋白质结构。
CASP 吸引了许多研究人员,但解决预测问题被证明非常困难。研究人员在比赛中输入的预测与实际结构之间的对应关系几乎没有改善。突破发生在 2018 年,当时一位棋坛大师、神经科学专家和人工智能先驱进入了该领域。
棋坛大师进入蛋白质“奥林匹克”
让我们快速了解一下 Demis Hassabis 的背景:他 4 岁开始下棋,13 岁时就达到了大师水平。在青少年时期,他开始从事程序员和成功的游戏开发者职业。他开始探索人工智能,并从事神经科学,在那里他做出了几项革命性的发现。他利用对大脑的了解来开发更好的 AI 模型。2010 年,他与人共同创立了 DeepMind,这是一家开发了流行棋盘游戏的人工智能模型的公司。该公司于 2014 年被谷歌收购,两年后,DeepMind 在全球范围内引起了关注,因为该公司实现了许多人当时认为的人工智能的圣杯:击败世界上最古老棋盘游戏之一的围棋冠军。
然而,对 Hassabis 来说,围棋不是目标,而是开发更好的人工智能模型的手段。取得这一胜利后,他的团队已准备好解决对人类来说更重要的问题,因此他们在 2018 年注册了第 13 届 CASP 竞赛。
AI 模型意外获胜
在前几年,研究人员为 CASP 预测的蛋白质结构,其准确率最高达到 40%。Hassabis 团队使用其 AI 模型 AlphaFold,达到了近 60%。他们获胜了,优异的结果让许多人感到意外——这是意外的进步,但解决方案仍然不够好。为了取得成功,预测的准确率必须与目标结构相比达到 90%。
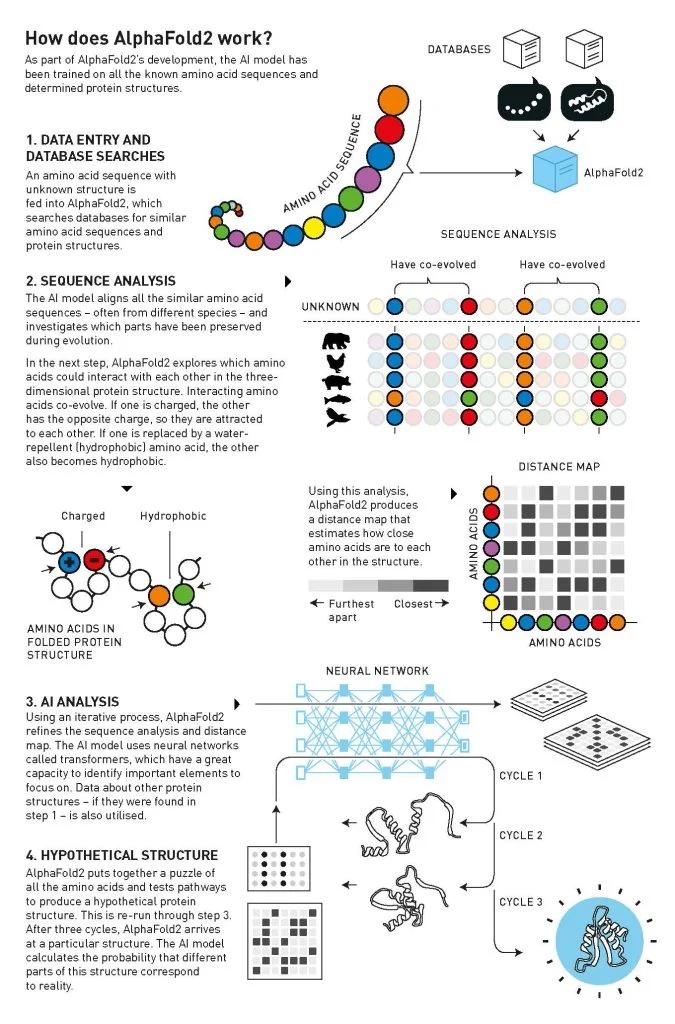
图 2. AlphaFold2 如何工作?(来源:Johan Jarnestad)
Hassabis 和他的团队继续开发 AlphaFold——但,不管他们多么努力,算法始终无法完全实现。残酷的事实是,他们已经走到了死胡同。团队疲惫不堪,但一位相对较新的员工对如何改进 AI 模型有决定性的想法:John Jumper。
John Jumper 迎接生物化学的巨大挑战
John Jumper 对宇宙的迷恋使他开始学习物理学和数学。然而,在 2008 年,当他开始在一家使用超级计算机模拟蛋白质及其动力学的公司工作时,他意识到物理知识可以帮助解决医学问题。
Jumper 在 2011 年开始攻读理论物理学博士学位时,将他对蛋白质的新兴趣带到了他的工作中。为了节省计算机容量(这在大学里非常短缺),他开始开发更简单、更巧妙的蛋白质动力学模拟方法。很快,他也接过了生物化学重大挑战的战旗。2017 年,他刚刚完成博士学位,就听说 DeepMind 在极度保密的情况下开始预测蛋白质结构。他向他们发送了一份求职申请。他对蛋白质模拟的经验意味着他对如何改进 AlphaFold 有创造性想法,因此,在团队开始原地踏步后,他得到了赏识。Jumper 和 Hassabis 共同领导了彻底改革 AI 模型的工作。
改进的人工智能模型取得惊人成果
新版本——AlphaFold2——体现了 Jumper 对蛋白质的了解。团队还开始使用最近人工智能取得巨大突破背后的创新:称为“transformers”的神经网络。这些网络能够以比以前更灵活的方式在大量数据中找到模式,并且可以有效地确定应该关注什么才能实现特定目标。
该团队在大规模已知蛋白质结构数据库和氨基酸序列(图 2)中训练 AlphaFold2,新的 AI 架构在第十四届 CASP 竞赛开始时就开始交付良好的结果。
2020 年,当 CASP 的组织者评估结果时,他们意识到生物化学 50 年的挑战已经结束。在大多数情况下,AlphaFold2 的表现几乎与 X 射线晶体学一样出色,这令人震惊。2020 年 12 月 4 日,CASP 的创始人之一 John Moult 在比赛结束时问道——接下来是什么?
我们将在后面回答这个问题。现在,我们将回到过去,关注 CASP 的另一位参与者。让我们介绍 2024 年诺贝尔化学奖的另一半,它涉及从零开始创建新蛋白质的艺术。
一本关于细胞的教科书使 David Baker 改变方向
当 David Baker 在哈佛大学开始学习时,他选择了哲学和社会科学。然而,在进化生物学课程中,他遇到了现在经典的教科书《细胞分子生物学》的第一版。这导致他改变了人生方向。他开始探索细胞生物学,最终他对蛋白质结构产生了浓厚的兴趣。1993 年,当他开始在华盛顿大学西雅图分校担任研究小组组长时,他接受了生物化学的重大挑战。他使用巧妙的实验开始探索蛋白质是如何折叠的。这为他提供了见解,他带着这些见解,在 20 世纪 90 年代末开始开发可以预测蛋白质结构的计算机软件:Rosetta。
Baker 于 1998 年使用 Rosetta 在 CASP 竞赛中首次亮相,与其他参赛者相比,它表现得非常好。这一成功导致了一个新的想法——David Baker 的团队可以使用 Rosetta 的反向功能。与其在 Rosetta 中输入氨基酸序列并输出蛋白质结构,他们应该能够输入所需的蛋白质结构并获得其氨基酸序列的建议,这将使他们能够创建全新的蛋白质。
Baker 成为蛋白质构造者
蛋白质设计领域——研究人员创建具有新功能的定制蛋白质——始于 20 世纪 90 年代末。在许多情况下,研究人员调整了现有的蛋白质,以便它们能够分解有害物质或作为化学制造行业中的工具。
然而,天然蛋白质的范围有限。为了增加获得具有全新功能的蛋白质的潜力,Baker 的研究小组希望从头开始创建它们。正如 Baker 所说,“如果你想建造一架飞机,你不应该从修改一只鸟开始;相反,你应该了解空气动力学的第一原理,并根据这些原理建造飞行器。”
一种独特的蛋白质问世
构建全新蛋白质的领域被称为从头设计(de novo design)。研究小组绘制了一种具有全新结构的蛋白质,然后让 Rosetta 计算哪种氨基酸序列可以生成所需的蛋白质。为此,Rosetta 搜索了所有已知蛋白质结构的数据库,并寻找与所需结构相似的蛋白质短片段。利用蛋白质能量分布的基础知识,Rosetta 对这些片段进行了优化,并提出了氨基酸序列。
为了研究软件的成功程度,Baker 的研究小组将建议的氨基酸序列的基因引入了细菌中,这些细菌产生了所需的蛋白质。然后他们使用 X 射线晶体学确定了蛋白质结构。
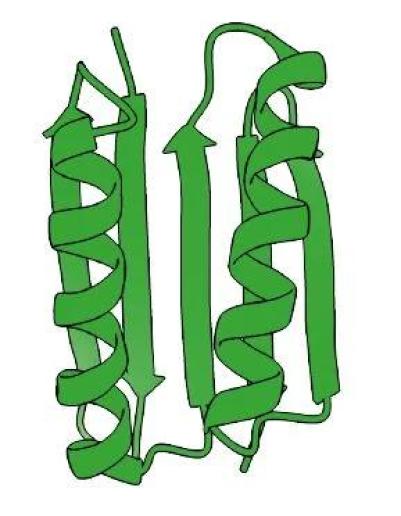
图 3. Top7 - 第一种与现有所有已知蛋白质完全不同的蛋白质。(来源:Terezia Kovalova)
结果表明,Rosetta 真的可以构建蛋白质。研究人员开发的蛋白质 Top7 几乎完全具有他们设计的结构。
Baker 实验室中令人瞩目的创造
Top7 对从事蛋白质设计的研究人员来说是一个惊喜。那些以前创建过从头开始设计的蛋白质的人只能模仿现有结构。Top7 的独特结构在自然界中不存在。此外,由 93 个氨基酸组成的蛋白质,比以前使用从头开始设计方法产生的任何蛋白质都要大。
Baker 于 2003 年发表了这项发现。这是令人惊叹的发展的第一步;Baker 实验室中涌现出了许多令人瞩目的蛋白质创造(图 4)。他还发布了 Rosetta 的代码,因此全球的研究团体都在继续开发该软件,寻找新的应用领域。
是时候为 2024 年诺贝尔化学奖画上圆满的句号了。现在怎么办?
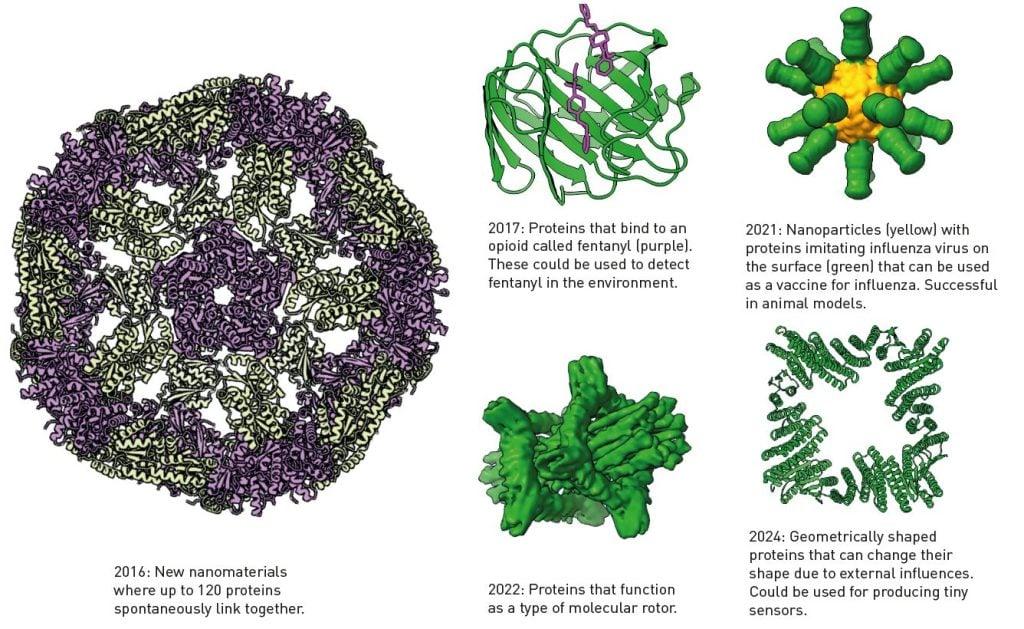
图 4. 使用 Baker 的 Rosetta 程序开发的蛋白质。(来源:Terezia Kovalova)
曾经需要花费数年时间的工作现在只需几分钟
当 Demis Hassabis 和 John Jumper 证实 AlphaFold2 真的起作用时,他们计算了所有人类蛋白质的结构。然后,他们预测了迄今为止研究人员在绘制地球生物图时发现的几乎所有 2 亿种蛋白质的结构。
DeepMind 还公开了 AlphaFold2 的代码,任何人都可以访问它。该 AI 模型已成为研究人员的金矿。到 2024 年 10 月,AlphaFold2 已被来自 190 个国家的 200 多万人使用。以前,获得蛋白质结构通常需要数年时间,现在只需几分钟就能完成。该 AI 模型并不完美,但它会估计它生成的结构的正确性,因此研究人员知道预测的可靠性如何。图 5 显示了 AlphaFold2 帮助研究人员的几个例子。
2020 年 CASP 竞赛结束后,当 David Baker 意识到基于 transformer 的 AI 模型的潜力时,他将其中一个添加到 Rosetta 中,这也有助于从头开始设计蛋白质。近年来,Baker 实验室一个接一个地涌现出了令人惊叹的蛋白质创造(图 4)。
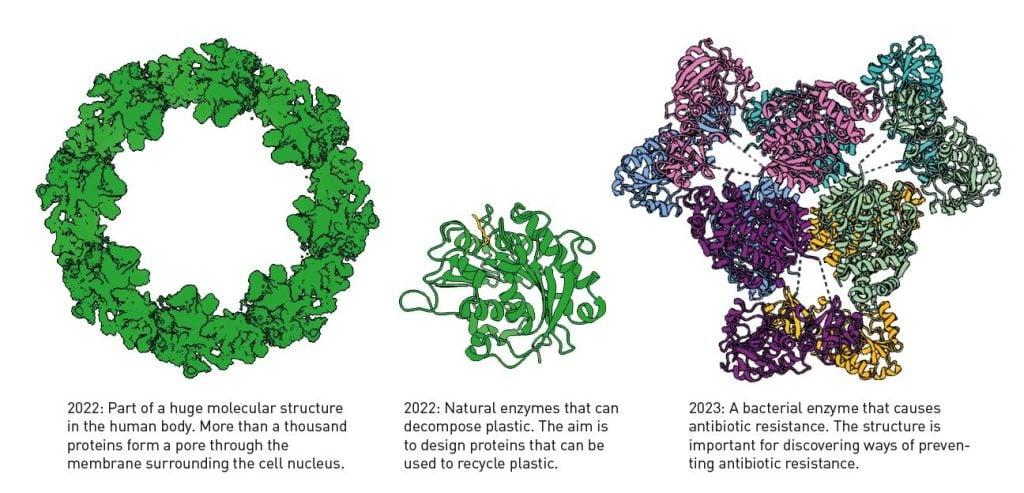
图 5. 使用 AlphaFold2 确定的蛋白质结构。(来源:Terezia Kovalova)
造福人类的飞速发展
蛋白质作为化学工具的惊人多功能性体现在生命的巨大多样性中。我们现在可以如此轻松地可视化这些小型分子机器的结构,这令人难以置信;这使我们能够更好地了解生命的运作方式,包括某些疾病是如何发展的、抗生素耐药性是如何发生的,以及某些微生物是如何分解塑料的。
能够创建具有新功能的蛋白质也同样令人惊叹。这可能导致新的纳米材料、靶向药物、疫苗的更快速开发、小型传感器和更环保的化学工业——这只是人类最受益的一些应用领域。
原文