10月9日,《细胞》杂志(Cell)刊发了中山大学与阿里云、悉尼大学教授Edward Holmes合作的最新科研成果。他们报告了全球范围的180个超群、16万余种的核糖核酸(RNA)病毒发现,这是迄今为止规模最大的RNA病毒研究,大幅扩展全球RNA病毒的多样性。
“新病毒的发现,刷新着科学家对病毒圈的认识。”论文共同通讯作者、中山大学医学院教授施莽对《中国科学报》表示,该研究将人工智能技术应用于病毒鉴定,发现了传统研究方法未能发现的病毒“暗物质”,为病毒学研究开创了一种全新的范式。
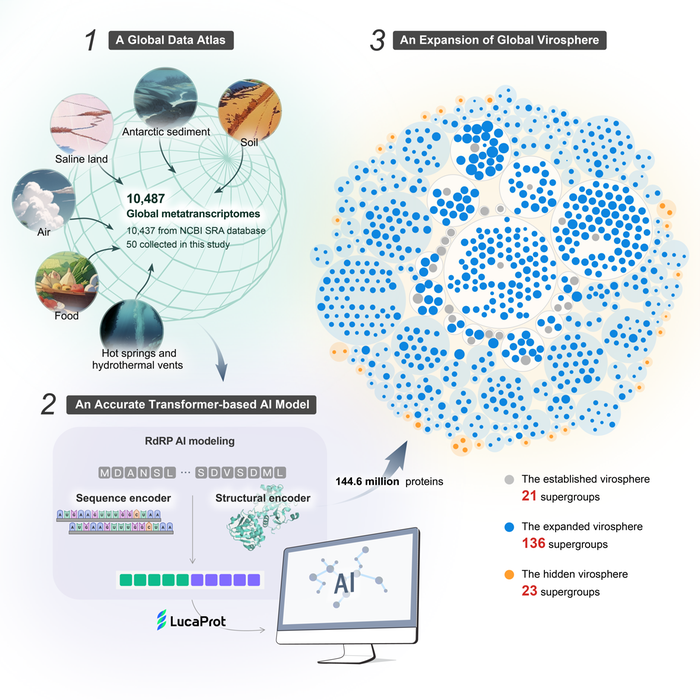
使用人工智能对全球病毒圈深度挖掘并分类。研究团队 供图
?
传统病毒发现方法遇瓶颈
病毒是世界上最神秘的微生物,与人类健康息息相关。全球病毒种类繁多且高度分化,在生态系统中扮演着至关重要的角色。但是,目前已知的病毒种类仍十分有限,用更高效、更精准的方法发现和鉴定新病毒,一直是病毒学研究的基础工作。
最早,人们通过分离培养病毒,在显微镜下观察确认病毒的存在。随着生命组学的发展,科学家们能够利用测序技术,比较未知病毒和已知病毒核酸序列的相似性,识别和鉴定新病毒。这种方法十分依赖对病毒的既有认知。
“传统的病毒发现方法包括病毒分离和生命组学的生物信息学分析,高度依赖既有知识。面对RNA病毒这种高度分化、种类繁多且容易变异的病毒识别效率低,尤其在面对缺乏同源性或同源性极低的‘暗物质病毒’时,这种序列同源性比对的方法很容易失灵。”施莽说。
人工智能技术的引入使突破“已知”寻找“未知”成为可能。研究中,团队采用的核心算法LucaProt是一种能够深度学习的Transformer模型,在大量学习病毒和非病毒基因组序列后,可以自主形成一套关于病毒的判断标准,从而在大量的RNA测序数据集中挖掘出病毒序列。
在测试中,LucaProt表现出极高的准确性和特异性,假阳性率为0.014%,假阴性率为1.72%。在与其他病毒挖掘工具的对比中,它也在处理较长序列的方面展现出优势。
“人工智能的算法模型能够挖掘出我们之前忽略或根本不知道的病毒,这种能力在疾病防控和新病原的快速识别中尤为重要。特别是在疫情暴发时,人工智能的速度和精度可以帮助科学家更快地锁定潜在病原体。”施莽说。
大幅拓宽RNA病毒库
利用LucaProt,研究团队对来自全球生物环境样本的10487份RNA测序数据进行病毒挖掘,发现了超过51万条病毒基因组,代表超过16万个潜在病毒种及180个RNA病毒超群,使RNA病毒超群数量扩容约9倍。其中23个超群无法通过序列同源方法识别,被称为病毒圈的“暗物质”。

新发现病毒的遗传多样性(黄色部分)。研究团队 供图
?
“这些病毒不仅指感染人类的病原体,还包括广泛存在于环境中的、感染各类生物的病毒。例如:各种动物、植物、单细胞原生生物、真菌、细菌和古菌都可能感染病毒。”论文第一作者、中山大学医学院博士侯新表示,深入了解环境中的病毒,有助于我们更好地理解整个生态系统的运作机制。“我们可以利用这种方法发现与人类疾病密切相关的病毒,用于新发传染病的监测和预警。”
依靠数据挖掘出来的新病毒,是否会停留在数字层面?侯新表示,通过对新发现病毒的分析,科学家们对病毒圈的认知也在不断深化。
通过进一步分析,研究团队发现迄今为止最长的RNA病毒基因组,长度达到47250个核苷酸;识别出超出以往认知的基因组结构,展示了RNA病毒基因组进化的灵活性;此外,在高温的深海热泉等极端环境中,RNA病毒依旧存在多样性。
“病毒的多样性远超人类想象,人类目前所看到的仍是冰山一角。”施莽表示,研究展示了病毒多样性的深度,但广度仍有待